La codificación genética de cualquier proteína no es uniforme en todos los organismos de una misma especie. De hecho, dicha información tiene al menos 10 000 cambios en el genoma codificante humano, aunque un 15% de ellos tienen el potencial de cambiar la función del producto proteico final 1. Si esto se pusiera en el contexto del estudio de la diversidad genética tanto del virus como del huésped podría mejorarse el diagnóstico, seguimiento y tratamiento de la infección por COVID-19 2, 3.
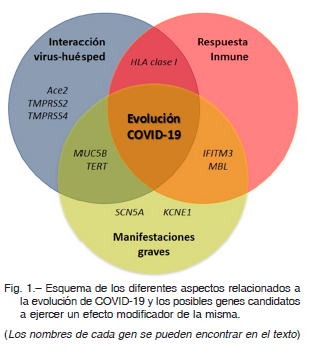
Aunque es poca la información disponible al respecto, sin duda será un área de intenso estudio, en especial en lo relacionado a las estructuras de reconocimiento entre el virus SARS-CoV-2 y la célula huésped, la respuesta de ésta y de cualquier otro tejido que intervenga en el desarrollo de los cuadros graves de la enfermedad (Fig. 1).
Variantes genéticas relacionadas a la interacción virus-célula huésped y de moléculas implicadas en la fisiología de COVID-19
La enzima conversora de angiotensina 2 (ACE2) es el receptor de entrada para el SARS-CoV-2. Su interacción con la proteína viral spike 2, 3 y con la proteasa transmembrane protease serine 2 (TMPRSS2) permite la internalización del virus. Un trabajo con enfermos italianos mostró que existen al menos cuatro variantes del gen ACE2 que pueden modificar dichas interacciones y explicar las diferencias en la velocidad de transmisión y la gravedad de COVID-19 encontradas en esa población 4. También se ha postulado que el efecto de variantes que exacerben o mitiguen la gravedad de COVID-19 sería más evidente en los hombres porque ACE2 se encuentra en el cromosoma X 5, 6. Sin embargo, un análisis bioinformático, también basado en población italiana, no puede corroborar estas aseveraciones sobre ACE2 y apunta a un desbalance en la distribución de alelos en diferentes variantes del gen TMPRSS2 como posible explicación de las diferencias en gravedad y prevalencia por sexo de la enfermedad 7.
Variantes genéticas relacionadas a la respuesta inmune en COVID-19
La experiencia adquirida luego de las epidemias previas por coronavirus (SARS-CoV y MERS-CoV), e incluso de la neumonía ocasionada por influenza, dejan claro que el cuadro de tormenta de citoquinas y quimiocinas liberadas por las células efectoras del sistema inmune juega un rol central en el devenir de COVID-19 5. El complejo mayor de histocompatibilidad es un conjunto de genes cuyos productos proteicos participan en la presentación de antígenos a los linfocitos T mediante la producción de antígenos leucocitarios que activan los procesos intracelulares de los linfocitos T para generar una respuesta inmunitaria. Este complejo se divide en dos grandes familias llamadas clase I y II, que codifican para proteínas intervinientes en la presentación de antígenos intra- y extra-celulares, respectivamente. El complejo mayor de histocompatibilidad contiene numerosas variantes genéticas y diversos trabajos mostraron que varios polimorfismos correlacionaron con la susceptibilidad a infección por SARS-CoV y MERS-CoV 5, 8, 9. Un estudio de ensayo de afinidad entre los componentes de HLA de clase I y el proteoma de SARS-CoV2 postula que la portación del alelo HLA-B*46:01 podría conferir vulnerabilidad a padecer COVID-19, mientras que aquellos con HLA-B*15:03 podrían poseer protección por inmunidad cruzada basada en células T10, pero dicha información no ha sido corroborada en poblaciones infectadas y libres de infección.
Sin embargo, algunos estudios sugieren que la susceptibilidad a la infección por SARS-CoV2 o la gravedad de la enfermedad estarían reguladas por algunas variantes genéticas del sistema de presentación de antígenos, como en el gen de lectina de unión a manosa (MBL) y de la proteína transmembrana inducida por interferón 3 (IFITM3), pero dichos hallazgos necesitan ser replicados y confirmados 8, 11.
Variantes genéticas asociadas a complicaciones cardíacas y COVID-19
La posibilidad de asociar complicaciones o manifestaciones clínicas graves a determinado perfil genético puede resultar interesante para definir el riesgo de cada paciente y personalizar su seguimiento y/o tratamiento. Este puede ser el caso de complicaciones de los tratamientos llamados “cócteles corona”, ya que pueden aumentar el riesgo de desarrollar el síndrome de QT prolongado inducido por fármacos que podría desencadenar torsades de pointes, y llevar a muerte súbita inducidas por fármacos de uso en COVID-19 como cloroquina/hidroxicloroquina con o sin azitromicina o lopinavir/ritonavir 5, 12. Es de notar que existe evidencia acumulada respecto a que variantes en canales iónicos relacionados con la contractibilidad cardíaca pueden tener un rol en estos cuadros, en especial los polimorfismos p.Asp85Asn en el gen KCNE1 y p.Ser1103Tyr en el gen SCN5A, que codifican para el miembro 1 de la subfamilia Q de los canales de potasio dependientes de voltaje y la subunidad alfa 5 del canal de sodio dependiente de voltaje, respectivamente. Los polimorfismos p.Asp85Asn-KCNE1 y p.Ser1103Tyr-SCN5A confieren diferente riesgo de prolongación del QT y muerte súbita 13 y los autores argumentan que la mayor frecuencia de la variante p.Tyr1103-SCN5A en los afroamericanos de EE.UU. (1 cada 13 vs. 1 cada 130 individuos de otro origen) podría explicar la casi duplicación de la tasa de mortalidad por COVID-19 encontrada en Estados Unidos de Norteamérica para esta población en particular 14.
Perspectivas
Dado que buena parte de la documentación comentada aquí incluye revistas y repositorios de trabajos aún sin revisión por pares, queda por determinar con certeza si existe algún efecto que pueda modificar cualquier característica de la infección por SARS-CoV2.
Sería interesante investigar la variación genética en la región intermedia o clase III del complejo de histocompatibilidad, cuyos genes codifican proteínas del complemento y citoquinas que son claves en el desarrollo de la respuesta inmune. Por ello, variantes en dichos genes pueden modificar la evolución de la enfermedad de manera directa o bien pueden explicar las diferencias encontradas en los alelos de las clases I o II.
Otra posibilidad aún no explorada sería evaluar la susceptibilidad genética para el desarrollo de fibrosis pulmonar. Existen estudios en los que variantes en genes como MUC5B – el gen que produce la mucina, el principal mucinógeno pulmonar, y TERT – un gen muy activo en los neumocitos que codifica la telomerasa y así preserva la integridad de los telómeros – podrían predisponer a cuadros de fibrosis pulmonar idiopática, fibrosis pulmonar relacionada a neumonía por hipersensibilidad y vasculitis por depósito de colágeno 5.
También sería interesante evaluar si existe alguna variabilidad genética relacionada con las manifestaciones intestinales (dolor abdominal, diarrea) presentes en alrededor del 50% de los infectados. Es interesante el caso de TMPRSS2, en especial luego de los elegantes estudios realizados por Zang y col., en donde se muestra que la infección de los enterocitos depende de la expresión de los productos proteicos de este y otro gen (TMPRSS4) estrechamente relacionado 15 (Fig. 1).
Por último, se requieren análisis específicos para revelar la relación entre las complicaciones graves de la progresión de la infección y los tratamientos planteados a través de estudios de secuenciación masiva del genoma de las diferentes variantes del virus y de los huéspedes, que evalúen estos y otros genes que intervengan en la fisiopatología de COVID-19 para luego corroborar los mecanismos subyacentes a través de, por ejemplo, ensayos in vitro y ex vivo.
Pablo J. Azurmendi
Instituto de Investigaciones Médicas Alfredo Lanari, Universidad de Buenos Aires, Argentina
e-mail: pabloazur@hotmail.com
1. Ng PC, Levy S, Huang J, et al. Genetic variation in an individual human exome. PLoS Genet 2008; 4: e1000160.
2. Hussain M, Jabeen N, Raza F, et al. Structural variations in human ACE2 may influence its binding with SARS-CoV-2 spike protein. J Med Virol 2020; 10.1002/jmv.25832.
3. Sheikh JA, Singh J, Singh H, et al. Emerging genetic diversity among clinical isolates of SARS-CoV-2: Lessons for today. Infect Genet Evol 2020; 84: 104330.
4. Renieri A, Benetti E, Tita R, et al. ACE2 variants underlie interindividual variability and susceptibility to COVID-19 in Italian population. medRxiv 2020. doi: https://doi.org/10.1101/2020.04.03.20047977.
5. von der Thüsen J, van der Eerden M. Histopathology and genetic susceptibility in COVID-19 pneumonia. Eur J Clin Invest 2020. doi: 10.1111/eci.13259.
6. Darbani B. The expression and polymorphism of entry machinery for COVID-19 in human: Juxtaposing population groups, gender, and different tissues. Int J Environ Res Public Health 2020; 17: E3433.
7. Asselta R, Paraboschi E, Mantovani A, Duga S. ACE2 and TMPRSS2 variants and expression as candidates to sex and country differences in COVID-19 severity in Italy.medRxiv 2020. doi: https://doi.org/10.1101/2020.03.30.20047878.
8. Li X, Geng M, Peng Y, Meng L, Lu S. Molecular immune pathogenesis and diagnosis of COVID-19. J Pharm Anal 2020; 10:102-8.
9. Forouzesh M, Rahimi, A. Valizadeh R, Dadashzadeh RMA. Clinical display, diagnostics and genetic implication of novel Coronavirus (COVID-19) epidemic. Eur Rev Med Pharmacol Sci 2020; 24: 4607-15.
10. Nguyen A, David JK, Maden SK, et al. Human leukocyte antigen susceptibility map for SARS-CoV-2. J Virol 2020; JVI.00510-20.
11. Thevarajan I, Nguyen THO, Koutsakos M, et al. Breadth of concomitant immune responses prior to patient recovery: A case report of non-severe COVID-19. Nat Med 2020; 26: 453-5.
12. Zaidel EJ, Wyss Quintana FS, Sosa Liprandi A, et al. Hidroxicloroquina. Mensajes desde la cardiología en tiempos de pandemia por coronavirus. Medicina (B Aires) 2020; 80: 271-4.
13. Giudicessi JR, Roden DM, Wilde AAM, Ackerman MJ. Classification and Reporting of potentially proarrhythmic common genetic variation in long QT Syndrome genetic testing. Circulation 2018; 137: 619-30.
14. Giudicessi JR, Roden DM, Wilde AAM, Ackerman MJ. Genetic susceptibility for COVID-19-associated sudden cardiac death in African Americans. Hear Rhythm 2020; S1547-5271(20): 30419-7.
15. Zang R, Castro MFG, McCune BT, et al. TMPRSS2 and TMPRSS4 mediate SARS-CoV-2 infection of human small intestinal enterocytes. bioRxiv 2020. doi: https://doi.org/10.1101/2020.04.21.054015.
