ROMINA CANZONERI, EZEQUIEL LACUNZA, MARTÍN C. ABBA
Centro de Investigaciones Inmunológicas Básicas y Aplicadas (CINIBA), Facultad de Ciencias Médicas, Universidad Nacional de La Plata, Buenos Aires, Argentina
Resumen La batalla contra el cáncer ha avanzado enormemente en los últimos treinta años y la tasa de supervivencia se ha duplicado, sin embargo aún es difícil alcanzar una cura generalizada. El desafío reside en que el cáncer no es una enfermedad única, se trata de decenas de manifestaciones diferentes incluso dentro de una misma localización tumoral. Para la biología de sistemas, cada tumor sólido es un sistema único caracterizado por su heterogeneidad celular, su interacción con el microambiente en el que crece y se desarrolla, y su capacidad de adaptarse y modificarlo. Los avances recientes en la comprensión de los mecanismos moleculares que subyacen al cáncer están transformando el diagnóstico y el tratamiento de la enfermedad. En este sentido, se ha desarrollado un conjunto creciente de tratamientos capaces de atacar con mayor eficiencia a un tumor específico dando paso a un nuevo paradigma: el de la medicina de precisión. La genómica y la bioinformática son dos ejes fundamentales en el desarrollo y aplicación de la medicina personalizada. Estas tecnologías generan datos masivos (Big Data) que requieren de herramientas analíticas y personal capacitado para su análisis, integración y transferencia de la información hacia los médicos especialistas. En esta presentación se describen los principales avances en genómica y bioinformática aplicados a la medicina de precisión así como sus perspectivas futuras, desafíos y problemáticas.
Palabras clave: medicina de precisión, genómica, bioinformática, Big Data
Abstract The battle against cancer has advanced tremendously in the last thirty years and the survival rate has doubled. However, it is still difficult to achieve a generalized cure. The challenge is that cancer is not a unique disease; it is about dozens of different manifestations, even within the same tumor location. For systems biology, each solid tumor is a unique system characterized by its cellular heterogeneity, its interaction with the microenvironment in which it grows and develops, and its ability to adapt and modify it. Recent advances in understanding the molecular mechanisms that underlie cancer are transforming the diagnosis and treatment of the disease. In this sense, a growing set of treatments capable of attacking a specific tumor with higher efficiency has been developed, defining a new paradigm: the precision medicine in oncology. Genomics and bioinformatics are two fundamental pillars in this applied field. These technologies generate massive data (Big Data) that require analytical tools and trained personnel for the analysis, integration and transfer of the information to physicians. This presentation describes the concepts of personalized medicine, Big Data, the main advances in genomics and bioinformatics as well as their future perspectives and challenges.
Key words: precision medicine, genomics, bioinformatics, Big Data
Dirección postal: Martín C. Abba, CINIBA, Facultad de Ciencias Médicas, UNLP, Calle 60 y 120 S/N, 1900 La Plata, Buenos Aires, Argentina
e-mail: mcabba@gmail.com
Si bien la lucha contra el cáncer ha avanzado enormemente en los últimos treinta años y la tasa de supervivencia se ha duplicado, la búsqueda de una cura definitiva aún es una especulación utópica. El desafío reside en que el cáncer no es una única enfermedad, sino que se trata de decenas de manifestaciones diferentes incluso dentro de una misma localización tumoral. Bajo la perspectiva de la biología de sistemas, cada tumor sólido es un sistema particular caracterizado por su heterogeneidad celular, el microambiente tumoral (tejido circundante, sistema inmune, etc.) y su capacidad de interacción, adaptación y/o modificación del mismo 1. Cada tumor constituye una entidad única y variable, debido a las diversas combinaciones de mutaciones y mecanismos epigenéticos que acontecen durante la transformación maligna en alguna de los millares de células que constituyen al tumor en sus estadios más tempranos. Y dado que estas células tumorales pueden sumar nuevas mutaciones y nuevas variaciones genéticas mientras el tumor crece y se desarrolla, puede alcanzarse una casi infinita cantidad de variaciones a nivel genómico 2. Por este motivo, tratar el cáncer se vuelve una tarea compleja, donde los oncólogos intentan detener un blanco móvil, impredecible, con gran capacidad adaptativa, por lo cual no puede existir una única solución válida para todos los casos. Si bien la cirugía, la radiación y la quimioterapia siguen siendo las principales armas contra el cáncer, los avances en la comprensión de los mecanismos moleculares están generando nuevos enfoques para el diagnóstico y tratamiento de la enfermedad. Tan solo en los últimos cinco años, se han desarrollado un centenar de nuevos tratamientos oncológicos para combatir los cánceres más agresivos.
Desde la edición genética hasta la inmunoterapia, el futuro del tratamiento del cáncer se enfoca en encontrar una solución especializada para cada problema individual 3.
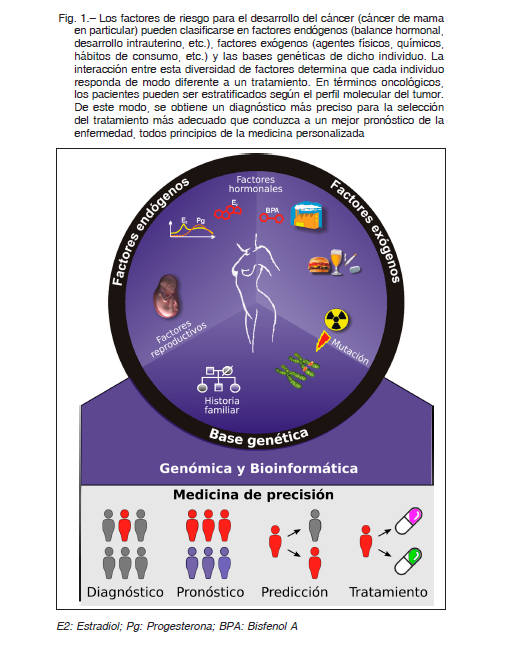
De esta manera, la medicina de precisión ofrece la posibilidad de ayudar a elegir tratamientos personalizados para atacar con mayor precisión a un tumor específico reduciendo los posibles efectos secundarios. El paradigma de la medicina de precisión no es nuevo, pero adelantos recientes en genómica y bioinformática han ayudado a materializar dicha disciplina (Fig. 1). De esta manera, la medicina personalizada supone grandes beneficios para los pacientes, lo cual permite a los oncólogos prescribir el tratamiento más adecuado desde una etapa temprana de la atención, minimizando el riesgo de que el paciente reaccione de manera adversa al medicamento, o bien, de que este último no surta efecto en su caso particular 4.
La medicina personalizada aumenta la eficiencia general de la asistencia sanitaria debido a que los perfiles moleculares de diagnóstico pueden descartar los tratamientos que no serán efectivos, evitando los costos que eso implica, e identificar el tratamiento con mayor posibilidad de éxito.
¿Cómo se identifican los cambios genéticos en el tumor?
La medicina de precisión es un modelo médico que propone la personalización de la atención médica a través del diagnóstico y las decisiones médicas asociadas (tratamientos y prácticas) que se adaptan al paciente individual4. Esta estrategia requiere, en primera instancia, determinar de manera precisa los mecanismos moleculares por los cuales un tumor prolifera, escapa al control del sistema inmune y/o resiste a su tratamiento. Los desarrollos tecnológicos-analíticos, computacionales y bioinformáticos experimentados durante la última década han permitido mover este modelo del terreno conceptual al pragmático. Por ejemplo, las actuales plataformas de secuenciación, inicialmente descritas como “secuenciadores de nueva generación” (NGS por sus siglas en inglés) permiten caracterizar de manera holística el epigenoma, el genoma y el transcriptoma de un tumor primario en particular. Inicialmente, se obtiene ADN y ARN de biopsias tisulares del tumor, o de biopsias liquidas: ADN y ARN circulantes o derivados de exosomas, provenientes del tumor. Luego se realizan los controles de calidad, para determinar la integridad y concentración de los ácidos nucleicos, antes de proceder a la construcción de las librerías en función de lo que se pretenda analizar [ejemplo: whole genome sequencing (WGSeq), whole exome sequencing (WESeq), TargetedSeq, MethylSeq, RNAseq, smallRNAseq, etc.]. Existen diversos secuenciadores, entre los cuales se pueden mencionar las plataformas de Illumina (series MySeq, HiSeq y NovaSeq) y Thermo-Fisher (series PGM, Ion Proton, Ion S5), entre otras4. Los archivos obtenidos de la secuenciación (archivos FASTQ) quedan disponibles para su descarga y posterior preprocesamiento (demultiplexing, control de calidad, recorte de adaptadores, etc.). Esta gran cantidad de datos debe ser procesada para la extracción de información no trivial como son la identificación de las vías de señalización por la cual las células tumorales proliferan o la presencia de variantes mutacionales clínicamente accionables (Fig. 2).
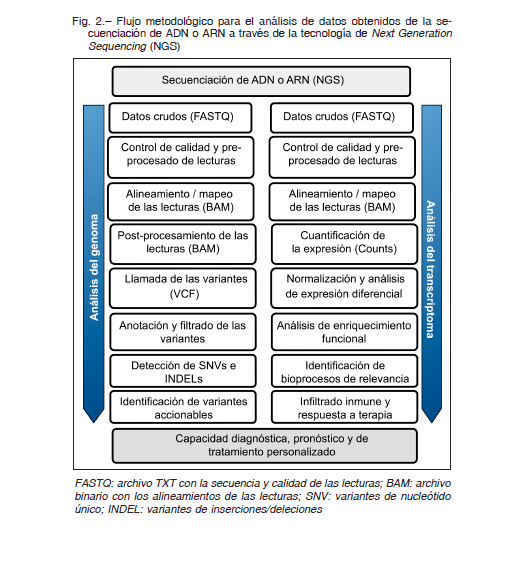
El proceso de secuenciación y análisis genera en consecuencia una cantidad relevante y compleja de datos que hacen que la bioinformática genómica se ubique dentro de las disciplinas que requieren estrategias procedimentales de Big Data 5. Si bien el avance tecnológico ha logrado que las tecnologías de NGS sean cada vez más accesibles en términos de disponibilidad, rapidez y costos, el actual cuello de botella reside en la capacidad de manejar un gran volumen de datos, así como en la capacidad de implementar, procesar computacionalmente e integrar los diversos flujos de trabajo que requiere cada aproximación experimental.
Dado que hay más de 3 millones de pares de bases en un exoma humano, distribuidos en 180 000 exones
(25 000 genes estructurales) 2, la secuenciación del exoma o transcriptoma completo para un proyecto pequeño de entre 10 y 30 muestras genera terabytes de datos crudos (archivos FASTQ) que deben ser pre-procesados y alineados en archivos BAM (la versión binaria de alineación de secuencia) que usualmente están en la escala de los gigabytes, dependiendo de la cobertura de secuenciación (el número promedio de veces que se lee cada base, profundidad de lectura) y la longitud de las lecturas5. La fase inicial de pre-procesamiento y alineamiento es la más demandante en cuanto a capacidad computacional, el resto de los procesos bioinformáticos requeridos varían radicalmente en función del tipo de estudio a realizar, pero son de tiempos de ejecución y capacidad de cómputo más moderados. En cualquier caso, una óptima infraestructura para Big Data facilita en gran medida el procesamiento y análisis de los datos.
La solución a los factores limitantes involucra un componente tecnológico y uno humano, que dependen de iniciativas e inversiones públicas y/o privadas. Diversos consorcios y/o compañías se encuentran desarrollando plataformas bioinformáticas en las nubes basadas en inteligencia artificial a fin de identificar y desarrollar flujos de trabajo más apropiados, e implementarlos en tiempo y forma con capacidad computacional a demanda. De esta manera se logrará acercar al usuario final (biólogos, genetistas, oncólogos) las herramientas analíticas necesarias para la apropiada interpretación de la información obtenida.
Inteligencia bioinformática colectiva
El término bioinformática ahora es ampliamente reconocido como un campo completo que abarca la biología, la medicina, la informática, las matemáticas, las estadísticas y la tecnología de la información. Las herramientas bioinformáticas (programas, aplicaciones, flujos de trabajo y bases de datos) constituyen un componente integral del proceso de investigación actual en ciencias biomédicas y otras áreas de la ciencia6. Desde la introducción de la tecnología de la información en la investigación biológica han surgido una gran cantidad de herramientas computacionales y bases de datos que han contribuido con importantes avances científicos. En este sentido, el advenimiento de las tecnologías de secuenciación de alto rendimiento ha estimulado el desarrollo de herramientas analíticas específicas en bioinformática genómica y requerido la implementación de flujos de trabajos altamente sofisticados.
En la última década, la noción de “datos biológicos” ha cambiado en magnitud y complejidad, desde un conjunto de cientos a conjuntos de millones de entidades (ejemplo: gen, variantes de splicing alternativo, proteína, isoformas, isla CpG, etc.). Este aumento exponencial en el volumen de datos biológicos ha estimulado el desarrollo de un número cada vez mayor de herramientas bioinformáticas 6.
Así como no hay una sola cura para el cáncer, tampoco existe una única herramienta para analizar los datos.
Ni bien se desarrollan nuevas herramientas y técnicas para procesar la información, surgen nuevas fuentes de datos que demandan mejoras radicales en los algoritmos de procesamiento y análisis. No menos relevante es la tangible falta de estándares en cuanto a la manera en que se recopilan, almacenan y procesan los datos genómicos, lo cual plantea evidentes problemáticas en la reproducibilidad de los resultados 5. Progresivamente, se ha adoptado un enfoque cooperativo que se manifiesta con el desarrollo de diferentes proyectos público-privados que democratizan tanto el uso de repositorios de datos genómicos como el de las herramientas y flujos de trabajo, reproducibles y de código abierto, entre los miembros de la comunidad científica global. Quizás uno de los mejores ejemplos es el proyecto Genomic Data Commons (GDC, NIH-NCI, EE. UU.) (https://portal.gdc.cancer.gov/) cuya misión es la de proveer a la comunidad científica un repositorio unificado que facilite el intercambio, la integración y el uso de los datos de los diversos proyectos de genómica funcional del cáncer, tales como el TCGA (The Cancer Genome Atlas), TARGET (Therapeutically Applicable Research to Generate Effective Treatments) y CGCI (Cancer Genome Characterization Initiative)7. De modo similar, los proyectos Bioconductor (https://www.bioconductor.org/) 8, Biocontainers (https://biocontainers.pro/)9 y Galaxy (https://usegalaxy.org/) 10 proveen herramientas bioinformáticas de código abierto desarrolladas por la comunidad científica así como biocontenedores y/o aplicaciones web que permiten su fácil y reproducible implementación. Por su parte, los desafíos DREAM son un esfuerzo científico abierto que apela a la colaboración participativa y al intercambio transparente de datos para evaluar herramientas analíticas existentes, sugerir mejoras y desarrollar nuevas soluciones (http://dreamchallenges.org/).
Desafíos que presenta la implementación de la medicina de precisión
Los sistemas sanitarios públicos y privados actuales están inmersos en un mar de datos: historias clínicas, resultados de ensayos clínicos, monitoreo de parámetros biométricos, imágenes diagnósticas diversas e información genética de pacientes. No solo se trata de un volumen creciente de datos, sino que buena parte son datos desestructurados y no pueden gestionarse con bases de datos tradicionales. Además, su manejo se complejiza por ser generados a gran velocidad lo cual es una dificultad no trivial para su integración, análisis y uso funcional. En este sentido, volumen, variedad y velocidad son precisamente las variables que caracterizan cualquier entorno de Big Data 5. Se trata de una disciplina en constante evolución debido al dinamismo de los avances tecnológicos y al constante desarrollo de nuevas herramientas analíticas.
Como todo advenimiento tecnológico, implica un período de “adaptación” por parte de la comunidad científica para su incorporación y un período aún mayor para su implementación en el ámbito médico. Las tecnologías son un componente vital para apoyar la medicina genómica, pero el volumen y la complejidad de los datos plantean desafíos para su uso en la práctica clínica. Esto, sumado a las barreras idiosincráticas institucionales, tecnológicas/humanas y legales hacen de la genómica clínica un desafío no menor a la hora de su implementación en el sistema sanitario.
Adecuación tecnológica y de profesional especializada
La tecnología incluida en los proyectos de Big Data ya es una realidad desde hace algún tiempo, y sus pilares fundamentales son los sistemas de archivos distribuidos, las bases de datos escalables, los programas de tratamiento masivo (tipo Hadoop), el cloud computing e Internet 5.
Pero esta tecnología tiene que consolidarse aún en el sector sanitario, por lo que será preciso que aumenten las inversiones públicas y privadas para este tipo de soluciones.
Cabe destacar que, quizá el factor más importante sea el de los recursos humanos: científicos idóneos y capacitados para el análisis e interpretación de los datos en el ámbito de la salud. Ellos son los responsables de brindar el soporte adecuado a los médicos en la toma de decisiones relativas a sus pacientes.
Salvaguardas legales y seguridad de los datos genómicos
Para que Big Data pueda entrar en escena y se consigan los mejores resultados, es preciso almacenar una ingente cantidad de datos procedentes de pacientes. Estos datos son extremadamente sensibles, siendo necesario garantizar el cumplimiento de la ley de protección de datos personales para garantizar su seguridad, confidencialidad e integridad. Las salvaguardas de la privacidad incluyen la utilización del cifrado de datos, la protección con contraseña, la transmisión segura de datos, el control de las metodologías de transferencia y el funcionamiento de estrategias institucionales contra los ataques de datos y el abuso malicioso de los mismos 11. Por ejemplo, los Principios de Prácticas Justas de Información (FIPP, sus siglas en inglés) ofrecen un marco para permitir el uso compartido y el uso de datos en base a las pautas adoptadas por el Departamento de Salud y Servicios Humanos de los EE.UU. 12.
Estado actual y perspectivas
La genómica y la bioinformática son fundamentales en el desarrollo y aplicación de la medicina personalizada de precisión en oncología en particular, y en la medicina del futuro en general. Estas tecnologías y herramientas analíticas deben implementarse de una manera integrada con la información disponible en las historias clínicas electrónicas y brindar metodologías interactivas de interpretación para facilitar su utilización por parte de los médicos especialistas. Todo ello debe lograrse en el más estricto marco de las salvaguardas éticas y de seguridad, por tratarse de información personal altamente sensible y sujeta de protección legal. Además, este cambio revolucionario en la forma de hacer medicina necesitará de la máxima implicación y colaboración de todos los actores del sector sanitario.
Gracias al estudio masivo de los datos se pueden determinar los porcentajes de curación de distintas terapias y así aplicar las más efectivas según las características individuales de cada paciente, reduciendo las posibilidades de que el tratamiento acabe en fracaso. Por ejemplo, el cáncer colorrectal (CCR) es una enfermedad molecular heterogénea, caracterizada por un patrón parcialmente definido de cambios moleculares que afectan varias vías moleculares. Esta diversidad de tumores ha desafiado las terapias desarrolladas durante los últimos años, lo que lleva a la necesidad de seleccionar grupos de pacientes con alteraciones moleculares similares, que pueden atenderse con terapias más personalizadas. En este sentido, un número creciente de ensayos clínicos se ha centrado en el uso de nuevos medicamentos dirigidos contra vías específicas para ser utilizados solos o en combinación 13. La correcta estratificación de los pacientes y la elección adecuada de los agentes terapéuticos conducirán eventualmente a avances significativos en el tratamiento del CCR y el cáncer en general. La heterogeneidad intratumoral, por otra parte, plantea desafíos aún más importantes. La coexistencia de múltiples subclones con diferentes conjuntos de alteraciones moleculares y diferentes sensibilidades a los medicamentos implica que las estrategias terapéuticas dirigidas a las alteraciones predominantes podrían no ser efectivas contra todo el tumor 14.
El advenimiento del single cell RNA sequencing, por ejemplo, es una de las metodologías que está generando una nueva masa de datos genómicos que requerirán ser analizados para descubrir nuevas entidades dentro de un mismo tumor y así ajustar cada vez más la precisión de las terapias. Queda por ver si la implementación a gran escala de este concepto es práctica y económicamente viable.
Las tecnologías de base genómica develan nuevos desafíos y afortunadamente evolucionan para enfrentarlos.
Aunque ya se están viendo los primeros avances, sobre todo en el diagnóstico y tratamiento del cáncer, queda mucho por hacer para llegar a una verdadera medicina personalizada de precisión.
Agradecimientos: Este trabajo ha sido financiado con el apoyo de los subsidios PICT2015-0149 (FONCyT, ANPCyT, Argentina) y 1U54CA221208-1 (NIH-NCI, EE.UU.)
Conflicto de intereses: Ninguno para declarar
Bibliografía
1. Faratian D, Bown JL, Smith VA, Langdon SP, Harrison DJ. Cancer systems biology. Methods Mol Biol 2010; 662: 245-63.
2. Quackenbush J. The Human Genome: Book of Essential Knowledge. Reino Unido: Imagine Publishing Inc., 2011.
3. Jaffee EM, Dang CV, Agus DB, et al. Future cancer research priorities in the USA: a Lancet Oncology Commission. Lancet Oncol 2017; 18: e653-706.
4. Krzyszczyk P, Acevedo A, Davidoff EJ, et al. The growing role of precision and personalized medicine for cancer treatment. Technology (Singap World Sci) 2018; 6: 79-100.
5. He KY, Ge D, He MM. Big data analytics for genomic medicine. Int J Mol Sci 2017; 18: pii: E412.
6. Levin CL, Dynomant E, Gonzalez BJ, et al. A data-supported history of bioinformatics tools. arXiv 2018; arXiv: 1807.06808.
7. Jensen MA, Ferretti V, Grossman RL, Staudt LM. The NCI Genomic Data Commons as an engine for precision
medicine. Blood 2017; 130: 453-9.
8. Huber W, Carey VJ, Gentleman R, et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nat Methods 2015; 12: 115-21.
9. da Veiga Leprevost F, Grüning BA, Alves Aflitos S, et al. BioContainers: an open-source and community-driven framework for software standardization. Bioinformatics 2017; 33: 2580-2.
10. Afgan E, Baker D, Batut B, et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Res 2018; 46: W537-44.
11. Middleton A. Society and personal genome data. Hum Mol Genet 2018; 27:R8-13.
12. Baker DB, Kaye J, Terry SF. Governance through privacy, fairness, and respect for individuals. EGEMS (Wash DC) 2016; 4: 1207.
13. Palma S, Zwenger AO, Croce MV, Abba MC, Lacunza E. From molecular biology to clinical trials: toward personalized colorectal cancer therapy. Clin Colorectal Cancer 2016; 15: 104-15.
14. Yates LR, Gerstung M, Knappskog S, et al. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nat Med 2015; 21: 751-9.
– – – –
[…] Y se debe dar un toque de atención para que la medicina por ganancia –cosa que no está del todo mal, sólo que ¿para quién la ganancia?– no implique exclusivamente la ganancia (honesta) de una empresa de financistas, inversores, economistas, gerentes y promotores excluyendo médicos y la atención adecuada. Esto es característicamente una exigencia ética y debiera complementarse con un compromiso médico profesional de no participar en empresas que explotan injustamente el trabajo médico, lo cual no es fácil, porque una mala remuneración es mejor que ninguna. ¿Hay algún lugar en este panorama para el hospital público? Debiéramos comprender que finalmente la atención médica la paga la gente y que hay quién no puede pagar. Este parece un argumento casi irrefutable en favor del hospital público si nos atenemos al derecho a la salud, pero hay otros argumentos además del humanitario: epidemiológicos, de educación médica y de política sanitaria. Y hay infinidad de actos médicos que no dan ganancia. El hospital público debiera proveer los adecuados niveles de complejidad y no explotar el trabajo gratuito de los médicos, hecho que parecería menos grave en estos casos porque no se trata de medicina por ganancia.
Samuel Finkielman (1932-2013)
En: Difícil para médicos (Editorial). Medicina (B Aires) 1998; 58: 327-8
