GLADYS E. CALDERÓN1, JULIA BRIGNONE1, MARÍA LAURA MARTIN1, FABIÁN CALLERI1, CARINA SEN1,
NATALIA CASAS3, ROGELIO CALLI4, ANABEL SINCHI1, DELIA ENRIA1, SILVANA LEVIS1
1Instituto Nacional de Enfermedades Virales Humanas (INEVH) Dr. Julio I. Maiztegui – ANLIS, Pergamino, 2SwissMedical, Medicina Privada, San Miguel de Tucumán, 3Programa Nacional de Control de Enfermedades Zoonóticas – Ministerio de Salud de la Nación, 4Dirección de Epidemiología – Ministerio de Salud Pública de la Provincia de Tucumán
Resumen Se describe un brote de síndrome pulmonar por hantavirus en el departamento de Burruyacú, provincia de Tucumán. La detección en 2016 de un caso de hantavirosis en una joven de 23 años –en ese momento considerado el primero ocurrido en dicha provincia– promovió un estudio epidemiológico exhaustivo, que permitió detectar retrospectivamente otro caso ocurrido en un niño de 5 años, un mes antes, en el mismo departamento. La infección fue confirmada por serología en ambos casos (caso 1 en muestras de 4 y 7 días de evolución, caso 2 en muestra a los 4 días). En ambos casos el genotipo viral fue caracterizado como HU39694 y los contactos fueron serológicamente negativos. En las áreas fueron identificados roedores pertenecientes a los géneros Akodon y Calomys y a la especie Mus musculus, pero no a Oligoryzomys, el reservorio habitual del genotipo HU39694. Tampoco se detectaron anticuerpos anti-hantavirus en suero de los roedores capturados. La ausencia de registro de viajes a área endémica de este genotipo y los hábitos recreacionales de los pacientes, sumados a los resultados serológicos negativos para hantavirus en los contactos, permiten inferir la posible exposición de los pacientes a fluidos de roedores infectados durante actividades recreativas o sociales al aire libre en sus respectivas áreas de residencia. En conclusión, se demuestra la circulación en Tucumán del genotipo viral HU39694, hasta ahora considerado restringido a la región pampeana central. Se extiende así a Tucumán el área endémica de hantavirosis, pero no se identificó el reservorio en el área.
Palabras clave: ARN de virus, genotipo viral, Sigmodontinae, síndrome de distrés respiratorio agudo, epidemiología
Abstract
Outbreak of hantavirus pulmonary syndrome in Tucumán, Argentina.
We describe an outbreak of hantavirus pulmonary syndrome in the Burruyacú Department, Province of Tucumán. The detection in 2016 of a case of hantavirosis affecting a 23-year-old woman, considered at that time to be the first case occurred in that province, promoted a thorough epidemiological study. The investigation allowed the retrospective detection of another case occurred one month earlier in a 5-year-old child in the same Department. In both cases, the infection was confirmed by serology (case 1 at days 4 and 7 of disease onset, case 2 at day 4) and the viral genotype was characterized as HU39694. The contacts of both cases were serologically negative for hantavirus. The rodents captured in the area belonged to genus Akodon, genus Calomys and species Mus musculus. Oligoryzomys, the known reservoir for this viral genotype, was not found. Specific anti-hantavirus antibodies were not detected in the captured rodents. Given that the patients had not visited hantavirus endemic areas and their contacts were negative for hantavirus, we infer that the patients were locally exposed to fluids of infected rodents during their usual social or recreational outdoor activities. In conclusion, we demonstrate that hantavirus HU39694 –a genotype until now considered to be restricted to the Central Pampas of the country– is circulating in the North Western province of Tucumán. The endemic area of hantavirosis is thus expanded to this province but the viral reservoir in the area has not yet been identified.
Key words: RNA viruses, viral genotype, Sigmodontinae, acute respiratory distress syndrome, epidemiology
Recibido: 29-XII-2017 Aceptado: 29-I-2018
Dirección postal: Gladys E. Calderón, Instituto Nacional de Enfermedades Virales Humanas Dr. Julio I. Maiztegui, Monteagudo 2510, 2700 Pergamino, Provincia de Buenos Aires, Argentina
e-mail: gcalderon@anlis.gov.ar
El síndrome pulmonar por hantavirus (SPH) es una enfermedad viral aguda causada por miembros del género Orthohantavirus, familia Hantaviridae. Puede presentarse como un síndrome febril inespecífico en su forma más leve o llegar hasta la manifestación más grave con una insuficiencia respiratoria aguda y shock cardiogénico. Presenta un período de incubación variable entre 4 y 42 días (rango medio de 9 a 12 días) con una evolución caracterizada por cuatro fases: prodrómica o febril, cardiopulmonar, diurética y de convalecencia1. Es una enfermedad con baja incidencia en Argentina, pero de relevancia para la salud pública por su relativamente alta letalidad (promedio 20%)2.
El genoma viral está constituido por tres segmentos de ácido ribonucleico (ARN) de cadena simple y polaridad negativa, denominados grande (L), mediano (M) y pequeño (S). Son virus transmitidos por roedores de la familia Cricetidae, subfamilias Murinae (fiebre hemorrágica con síndrome renal) y Sigmodontinae (síndrome pulmonar por hantavirus) que actúan como reservorios naturales. La infección de los roedores es asintomática, crónica, con eliminación persistente de virus, principalmente por saliva y orina. La transmisión a los humanos se produce fundamentalmente por la inhalación de aerosoles conteniendo partículas virales provenientes de las secreciones y excreciones de los roedores infectados. Otras posibles vías de transmisión son a través de la mucosa conjuntival, nasal o bucal o por mordedura de roedores infectados. La transmisión interhumana es excepcional y ha sido reconocida únicamente para el genotipo Andes, en la región sur del país3.
En Argentina, los primeros casos de SPH fueron confirmados mediante un estudio retrospectivo de muestras de pacientes notificados como Fiebre Hemorrágica Argentina en las provincias de Buenos Aires y Santa Fe4 y de enfermos con síndrome de distrés respiratorio agudo en la provincia de Salta (J Cortés y col., Congreso Interamericano de Infectología, 1994).
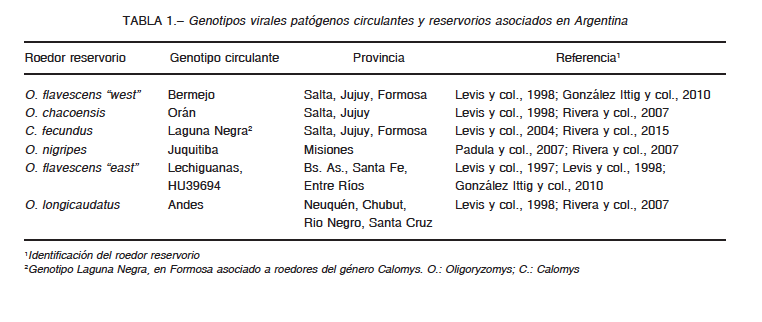
La confirmación de laboratorio de los casos se realiza por uno o más de los siguientes criterios: serología por ELISA (IgM o seroconversión IgG), detección de ARN viral mediante RT-PCR en coágulo u órgano, inmunohistoquímica en órganos de autopsia5. A la fecha, se han confirmado por diagnóstico de laboratorio 1550 casos humanos de SPH en Argentina, con mayor prevalencia en varones de 15 a 65 años de edad; aunque también se han registrado casos pediátricos y en mayores de 65 años6. Por otro lado, se han caracterizado e identificado los genotipos virales patógenos para el humano (Andes, Lechiguanas, HU39694, Juquitiba, Bermejo y Orán) que tienen como reservorio diferentes especies del género Oligoryzomys de las regiones sur, centro, noreste y noroeste de Argentina, además del genotipo Laguna Negra cuyo reservorio es Calomys fecundus (Tabla 17-13; N Pini y col. I Congreso Panamericano. V Congreso Argentino. II Congreso Bonaerense de Zoonosis 2006; C Sen y col. XII Congreso Argentino de Virología, 2017).
En 2016 se detectó un brote de SPH en la provincia de Tucumán, la cual se encontraba fuera del área endémica reconocida de la enfermedad. El objetivo del presente trabajo es describir los casos clínicos y los resultados de los estudios ecoepidemiológicos realizados en relación a dicho brote.
Materiales y métodos
El Caso 1 era una mujer de 23 años de edad, con nivel de educación superior y antecedentes de síndrome antifosfolipídico. Consultó el 29 de marzo de 2016 en un centro de atención primaria de la localidad de Yerba Buena, departamento del mismo nombre, provincia de Tucumán, su lugar de residencia, por un cuadro febril de 72 horas de evolución acompañado de cefalea, mialgias, náuseas, vómitos y dolor abdominal, sin antecedente de viaje. Al día siguiente, ingresó a unidad de terapia intensiva de un sanatorio privado, por persistencia del cuadro febril y alteración de la función respiratoria. Al 5to. día del comienzo de los síntomas, presentó deterioro del estado general y la tomografía de tórax mostró infiltrado pulmonar asociado a imágenes nodulares, lo que indujo la primera sospecha de SPH. Se inició tratamiento con piperacilina-tazobactam, claritromicina y oseltamivir. La paciente fue luego derivada a un establecimiento de la Ciudad Autónoma de Buenos Aires, a cuya unidad de terapia intensiva ingresó hemodinámicamente inestable, con parámetros de distrés respiratorio y con necesidad de vasopresores, asistencia respiratoria mecánica y oxigenación por membrana extracorpórea. El 2 de abril se practicó esternotomía de urgencia por taponamiento cardíaco y la paciente desarrolló falla multiorgánica. Los resultados del laboratorio clínico presentaron valores compatibles con un cuadro de SPH: hematocrito 56%, 80 100 glóbulos blancos/mm3, 47 000 plaquetas/mm3. Falleció el 6 de abril.
El caso 2 era un varón de 5 años de edad de la localidad de El Chañar, departamento de Burruyacú, provincia de Tucumán; previamente sano y sin antecedentes de viaje. Ingresó el 7 de febrero de 2016 a la guardia de hospital por síndrome febril de 24 horas de evolución, donde se indicó tratamiento
ambulatorio y pautas de alarma. El 9 de febrero fue traído nuevamente a la guardia, en consulta por persistencia del cuadro febril, vómitos y deterioro del estado general. Ese mismo día se lo ingresó a unidad de terapia intensiva del Hospital de Niños por deshidratación grave, insuficiencia respiratoria y diagnóstico presuntivo de shock séptico con foco pulmonar y falla multiorgánica. La radiografía de tórax mostró infiltrados algodonosos bilaterales. Los resultados de laboratorio presentaron un valor de 50% de hematocrito, 42 000 plaquetas/mm3, 26 600 glóbulos blancos/mm3. Se inició tratamiento con ceftriaxona y vancomicina. Falleció el mismo día.
Se obtuvieron 26 muestras de suero de los contactos del caso 1 (padres, hermanos, novio, hermanos políticos y amigos) y una muestra de la hermana del caso 2.
La investigación de anticuerpos específicos para hantavirus14 en suero de pacientes (IgM) y contactos (IgM e IgG) se realizó mediante la técnica de inmunoensayo ligado a enzima (ELISA). El antígeno viral utilizado en la prueba fue obtenido en cultivos celulares línea Vero E6 infectados con el genotipo Maciel, irradiado por radiación gamma. Los conjugados utilizados fueron anti-IgM y anti-IgG humanas marcadas con peroxidasa. Una muestra se consideró positiva si el título fue ≥ 1:400.
Con el objetivo de detectar e identificar el genoma viral, se realizó la extracción del ARN total de las muestras de suero de los pacientes al 4to.día de evolución de la enfermedad y RT-PCR anidada. La extracción del ARN se realizó utilizando el kit Qiamp Viral RNA Mini Kit (Qiagen Inc., Germany). La amplificación del genoma se realizó mediante RT-PCR anidada utilizando oligonucleótidos genéricos para hantavirus que amplifican fragmentos de la nucleoproteína del segmento S (447 nt) y de la región codificante de la glicoproteína G2 del segmento genómico M (513 nt).
La reacción de RT-PCR anidada se realizó utilizando un buffer 10X libre de Mg++ suplementado con MgCl2 1.8 mM, dNTPs 0.2 mM, 1.2 U de ADN polimerasa Platinum®Taq DNA Polymerase (Invitrogen, USA), en un volumen total de 30 μl. Para el primer ciclo de amplificación se utilizó una dilución 10-1 de retrotrancriptasa Super Script III Reverse Transcriptase (Invitrogen, USA). Para el segundo ciclo de amplificación se utilizó 3% del producto obtenido en el primer ciclo sin agregado de retrotranscriptasa.
Los productos de amplificación fueron utilizados para la secuenciación del genoma viral. Las bandas obtenidas fueron purificadas utilizando el kit QIAquick Gel Extraction Kit (Qiagen Inc., Germany) siguiendo las recomendaciones del fabricante y secuenciados con un equipo 3100 Avant Genetic Analyzer (Applied Biosystems).
Para los análisis filogenéticos, los alineamientos de las secuencias de distintos genotipos de hantavirus fueron realizados con la herramienta ClustalW implementada en el editor de alineamientos de secuencias BioEdit 7.2.015. La determinación de los modelos evolutivos se realizó con el programa JModelTest 2.1.416. Los análisis filogenéticos de los fragmentos S y G2 amplificados fueron realizados empleando el algoritmo MCMC (Metropolis-coupled Markov chain) en el programa MrBayes 3.5.217.
La investigación eco-epidemiológica –pilar fundamental de los estudios de brote en enfermedades zoonóticas– consistió en: (i) el análisis de la información epidemiológica registrada en la ficha de notificación de síndrome febril agudo inespecífico; (ii) la investigación de las actividades realizadas y los sitios frecuentados por los pacientes durante el período probable de contagio, a través de entrevistas con los familiares y otros contactos. Estas investigaciones permiten identificar probables sitios de contagio y establecer áreas de captura de roedores.
El caso 1 se trasladó en enero de 2016 desde su vivienda permanente, ubicada en zona residencial de la ciudad de Tucumán, a una finca ubicada en zona rural de la localidad de Marino (Departamento Burruyacú) donde permaneció durante el periodo estival. La finca, cuya principal actividad económica es la agricultura, presenta construcciones edilicias de calidad y mantenimiento excelentes. El peridomicilio está compuesto por más de 50 metros de parque alrededor de la vivienda, donde se encuentra una represa con cría de peces para actividades recreativas.
El caso 2 residía en El Chañar (Departamento Burruyacú), con una población de menos de 5000 habitantes. El niño vivía con sus padres en una casa de material, ubicada en el área periférica del pueblo, rodeada de campos dedicados a la agricultura. Los domicilios de otros familiares del menor, a cuyo cuidado eventualmente quedaba, estaban ubicados en el mismo sector del pueblo.
Con la información obtenida, se procedió a la selección de hábitats para el estudio de micro-roedores. A su vez, se evaluaron los probables sitios de contagio considerando la importancia del peridomicilio en la transmisión de los hantavirus al hombre18 y la preferencia de hábitat de los roedores reservorios de hantavirus en la Argentina.
El mayor esfuerzo de captura se realizó en el caso 1 por ser temporalmente el más cercano a la realización del muestreo. En este caso, se trabajó en la finca ubicada en el área rural de la localidad de Marino, donde se muestreó el domicilio y peridomicilio; en este último se colocaron trampas en los bordes de la huerta, bordes de acequia, bordes de represa, lotes con cultivos de caña forrajera y con plantación de nogales pecan.
En la localidad El Chañar fueron colocadas trampas en el domicilio de residencia y el peridomicilio del caso 2 y en el de sus familiares. Dentro de los peridomicilios se muestreó en lotes con malezas, cultivos de soja y caña de azúcar.
En la localidad de El Naranjo fueron colocadas trampas en dos lotes de cítricos lindantes con zona de selva nublada, sin relación con los casos.
Entre el 25 y el 28 de abril de 2016, se colocaron un total de 877 trampas de captura viva de roedores tipo Sherman cada 5 m en los sitios descriptos previamente. El cebo utilizado consistió en una mezcla de avena arrollada y pasta de maní. Las trampas se colocaron al atardecer y se revisaron por la mañana. Las capturas obtenidas fueron trasladadas a un laboratorio de campo siguiendo los procedimientos de bioseguridad establecidos19. El procesamiento de los roedores se realizó previa anestesia profunda con isofluorano y se obtuvo una muestra de sangre del seno retro-orbital. La conservación y traslado de las muestras se realizó en termos de nitrógeno líquido hasta su arribo al INEVH donde fueron conservadas a -70 °C.
Se realizó un análisis a campo de las características morfométricas de los roedores capturados para la identificación preliminar de género/especie. Entre los roedores capturados, para el análisis genético de especie fueron seleccionados los del género Calomys por ser reservorio de otro hantavirus en el país. La caracterización precisa de especie se realizó mediante el análisis del ADN mitocondrial (ADNm). La extracción del ADN total se realizó a partir de una muestra de su cola utilizando el kit DNeasy Blood and Tissue kit (Qiagen Inc., Germany). La amplificación del gen que codifica para el citocromo b del ADN mitocondrial se realizó mediante PCR utilizando los primers Mus14095/Mus15398 que amplifican un fragmento de 1140 nucleótidos. Los productos amplificados se visualizaron mediante electroforesis en gel de agarosa al 1%. La purificación y la secuenciación del ADN se realizaron utilizando el mismo kit y equipo de secuenciación descripto más arriba.
Los análisis filogenéticos se realizaron con el programa PAUP* 4.0b1020. Se realizaron búsquedas heurísticas para hallar los árboles de máxima parsimonia. Los soportes de los nodos fueron estimados mediante el método de bootstrap con 1000 réplicas, para obtener un árbol consenso de mayoría. Se utilizó una matriz y secuencias de referencia para la identificación de especímenes del género Calomys.
La búsqueda de IgG en la muestra de sangre de los roedores capturados se realizó mediante ELISA, aplicando la misma metodología descripta para las muestras de humanos, pero reemplazando el conjugado anti-IgG humana por uno anti IgG de ratón.
Resultados
La confirmación diagnóstica de los casos se obtuvo mediante la detección de anticuerpos de tipo IgM específicos para hantavirus con un título > 1:6400 en las muestras de 4 y 7 días (caso 1) y 4 días (caso 2) de evolución desde el comienzo de los síntomas.
La búsqueda de anticuerpos IgM e IgG por ELISA en las muestras de contactos de los casos estudiados, resultaron negativos.
Para los análisis filogenéticos del virus se incluyeron secuencias nucleotídicas del genotipo HU39694 obtenidas de humanos y roedores provenientes de diferentes localidades de la provincia de Buenos Aires, caracterizadas en nuestro laboratorio, y secuencias de otros genotipos de hantavirus circulantes en Argentina y en otros países de América depositados en el GenBank. Los árboles obtenidos con el método neighbor-joining con inferencia bayesiana mostraron topologías semejantes.
Las Figuras 1 y 2 muestran las reconstrucciones filogenéticas obtenidas por comparación de las secuencias nucleotídicas parciales de un fragmento de 377 nucleótidos para la NP (segmento S) y de un fragmento de 283 nucleótidos para la G2 (segmento M) mediante inferencia bayesiana. Los valores de probabilidad posterior (PP) se encuentran detallados en los árboles filogenéticos. Las secuencias del fragmento S y M de los genotipos Hu93659 (caso 1) y Hu91578 (caso 2), descriptas en este trabajo, presentaron una elevada similitud entre sí (99.5% y 99.3% para los segmentos S y M, respectivamente). Ambas resultaron formar un grupo bien definido con las cepas del genotipo HU39694 de la provincia de Buenos Aires (PP 0.99) y relacionado filogenéticamente con los genotipos LEC Y BMJ (PP 0.76). Resultados similares han sido descriptos previamente6. La comparación de las secuencias nucleotídicas de los genotipos circulantes en la provincia de Buenos Aires con las de Tucumán mostró una similitud del 96.3%-98.7% para el segmento S y 95.4%-97.5% para el segmento M. Las secuencias nucleotídicas del virus de los pacientes de Tucumán, caracterizadas como pertenecientes al genotipo HU39694, presentaron una variación
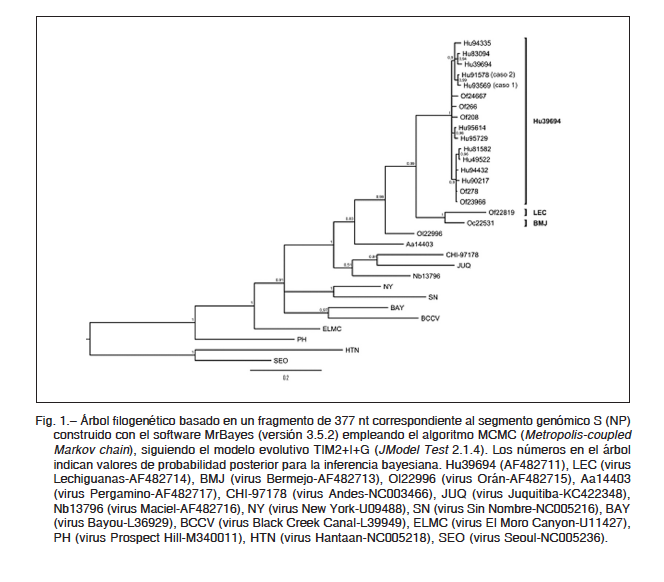

del 84.4-85.4% para el segmento S y 83.4%-84.5% para el segmento M con respecto al genotipo BMJV,
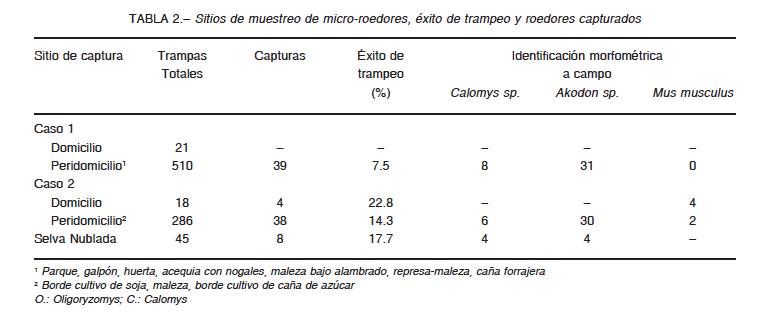
La captura de roedores se realizó durante tres noches con 847 trampas activas y se capturaron un total de 89 individuos, lo que representó un éxito de trampeo de 10.5%. El 93.2% de los animales capturados correspondió a la subfamilia Sigmodontinae y el 6.7% a la familia Muridae. El 74.1% correspondió al género Akodon, el 19.1% al género Calomys y el 6.7% a la especie Mus musculus; esta última se capturó solamente en relación al caso 2 (Tabla 2). En ambos casos, el mayor número de capturas se obtuvo en ambientes aledaños a cultivos de caña de azúcar y forrajera, que era parte del peridomicilio.
Los estudios del ADN mitocondrial en el género Calomys permitieron identificar a C. fecundus y C. musculinus.
Las muestras de sangre (n = 89) de los animales capturados resultaron negativas para la detección de anticuerpos totales específicos para hantavirus.
Discusión
Los resultados del presente trabajo describen la detección y la investigación de un brote de SPH en la provincia de Tucumán. La confirmación del brote resultó en la ampliación del área endémica de hantavirus en la región noroeste de Argentina. La confirmación diagnóstica de los casos se realizó mediante la detección de anticuerpos de tipo IgM por ELISA, los cuales aparecen en el período agudo y pueden perdurar hasta 6 meses. En el caso 1, dos muestras (día 4 y día 7) presentaron IgM positiva, a diferencia de los resultados obtenidos por otro laboratorio en una publicación anterior21.
La confirmación diagnóstica del caso 1 condujo a la búsqueda activa de casos, lo que permitió identificar y confirmar retrospectivamente el caso 2, ocurrido un mes y medio antes. La presentación clínica de ambos fue compatible con el SPH. La investigación epidemiológica mostró un patrón diferente en relación a la edad de los casos, el nivel socioeconómico, las condiciones de sanidad ambiental de las viviendas y los sitios que frecuentaban los pacientes. En ninguno de los dos casos pudo establecerse el antecedente epidemiológico indicativo de contacto con roedores o con sus excretas.
Estudios realizados por el INEVH17 demostraron la importancia del peridomicilio como sitio de riesgo de adquirir el SPH en Argentina; en tal sentido, y en base a la información epidemiológica obtenida en relación al caso 2 (pediátrico), consideramos que, de los sitios que habitualmente frecuentaba el niño, el peridomicilio sería el sitio probable de contagio
El análisis de la información epidemiológica del caso 1 permitió descartar una infección en áreas endémicas del SPH porque la paciente no viajó fuera de su provincia. Por otra parte, sus contactos resultaron serológicamente negativos, por lo que la conjetura formulada recientemente de contagio interhumano21 resulta improbable. Aunque tampoco encontramos evidencia de infección en los roedores capturados en el área, la hipótesis más plausible es que la paciente haya estado expuesta a fluidos de reservorios durante el desarrollo de sus habituales actividades deportivas y recreativas en ambientes silvestres.
La RT-PCR permitió detectar ARN viral en la muestra de suero del 4to día en ambos pacientes. El análisis filogenético de las secuencias de ARN amplificadas permitió caracterizarlas como pertenecientes al genotipo HU39694, el cual forma un grupo bien definido con Lechiguanas y Bermejo. Así queda demostrada la circulación del genotipo HU39694 en la provincia de Tucumán por la confirmación diagnóstica y caracterización del mismo genotipo en ambos casos.
En el sistema actual de nomenclatura de los hantavirus existen discrepancias en la denominación de los genotipos circulantes en Argentina. En esta publicación, se ha mantenido la denominación de genotipo HU39694 otorgada cuando se reconoció y caracterizó por primera vez este genotipo viral13. A pesar de las discrepancias en las nomenclaturas, el genotipo viral caracterizado en este trabajo agrupa con uno de los genotipos circulantes en la provincia de Buenos Aires, tal como observaron recientemente otros autores en muestras del caso 121.
La identificación inequívoca de las especies del género Oligoryzomys, reservorios de diferentes hantavirus, ha presentado cierta complejidad. Recientes estudios8, 9, 22, utilizando la secuenciación nucleotídica del gen citB, han permitido caracterizar las especies de Oligoryzomys y los hantavirus que transmiten. Levis et al.10 describieron la asociación de O. flavescens con el hantavirus genotipo HU39694 de la región centro del país. Recientes publicaciones describen diferencias genéticas entre las poblaciones de O. flavescens del este y del oeste del área de su distribución geográfica, las que dieron lugar a la denominación de subespecies (O. flavescens east y O. flavescens west)22. A la luz de los resultados del presente trabajo – la circulación de hantavirus genotipo HU39694 en la provincia de Tucumán y la ausencia de capturas de roedores del género Oligoryzomys – resulta indispensable continuar realizando capturas de roedores en la zona de estudio a fin de determinar el reservorio, dado que la distribución geográfica de la subespecie O. flavescens east, que ha sido caracterizada como reservorio de HU39694 en la zona centro de nuestro país, no alcanza la provincia de Tucumán.
La ocurrencia de los dos casos de infección por hantavirus en el departamento Burruyacú, en un período de dos meses podría estar relacionada a factores ecológicos, antrópicos y/o climáticos que conducen a la modificación de la dinámica de las poblaciones de roedores.
Los resultados de estas investigaciones fueron comunicados a las autoridades y a los agentes comunitarios. Se compartió información a través de medios masivos de comunicación y se procedió a capacitar al personal de salud sobre los diferentes aspectos de los hantavirus: mecanismo de transmisión, medidas de prevención, especies de roedores involucradas en la transmisión y diagnóstico de laboratorio. Este tipo de interacción adquiere particular relevancia porque se trata de una enfermedad emergente en una determinada región. Como además es una zoonosis transmitida por animales silvestres, la erradicación es virtualmente imposible. Por lo tanto, es fundamental que los organismos públicos y la población en general se involucren en acciones de prevención.
Finalmente, desde el punto de vista clínico, el SPH constituye una amenaza latente para la salud. Es imprescindible la sospecha temprana de esta enfermedad dentro de los síndromes febriles y la recolección de la información epidemiológica, no solo en las áreas endémicas sino también en aquellas áreas donde no se han registrado aún casos humanos. A la vez, la confirmación diagnóstica temprana de los casos contribuye al estudio de la epidemiología del SPH considerando que esta enfermedad presenta un patrón dinámico asociado a factores ambientales, laborales, recreativos, socio-económicos y culturales.
Agradecimientos: Al Dr. Jorge San Juan, Director Nacional de Epidemiología y la Dra. Norma Cudmani por el apoyo brindado; a la Licenciada Silvina Ortega y al Médico Veterinario Fernando Hilal por el apoyo en la búsqueda de información epidemiológica y la logística para las actividades en terreno. A Germán O´Duyer, José Paura y César Polidoro por el apoyo técnico para la captura y procesamiento de roedores silvestres y a Débora Zeljkovich por el apoyo técnico en el diagnóstico serológico.
Conflicto de intereses: Ninguno para declarar
Bibliografía
1. Enria D, Lázaro M, Levis S. Capítulo 35. Hantavirus. En: Virología Médica 4° Edición. Eds: G Carballal G y J Oubiña. CORPUS, Ciudad Autónoma de Buenos Aires, 2015; pp 623-6.
2. Ministerio de Salud. Presidencia de la Nación. Enfermedades Infecciosas. Hantavirus: Diagnóstico de Hantavirus: Guía para el equipo de salud 2016. En: http://www.msal.gob.ar/images/stories/bes/graficos/0000000065cnt-2016-guia-medica-hantavirus.pdf; consultado noviembre 2017.
3. Enria D, Padula P, Segura E, et al. Hantavirus pulmonary syndrome in Argentina. Possibility of person to person transmission. Medicina (B Aires) 1996; 56: 709-11.
4. Parisi M, Enria D, Pini N, Sabattini M. Detección retrospectiva de infecciones clínicas por Hantavirus en la República Argentina. Medicina (B Aires) 1996; 56: 1-13.
5. Ministerio de Salud. Presidencia de la Nación. Manual de Normas y Procedimientos de Vigilancia y Control de Enfermedades de Notificación Obligatoria. Revisión Nacional 2007. En: http://www.snvs.msal.gov.ar/descargas/Manual%20de%20Normas%20y%20Procedimientos%202007.pdf; consultado noviembre 2017.
6. Ministerio de Salud. Presidencia de la Nación. Sistema Nacional de Vigilancia en Salud, Sistema Nacional de Vigilancia Epidemiológica. En: http://www.msal.gob.ar/images/stories/boletines/Boletin-Integrado-De-Vigilancia-N322-SE32.pdf ; consultado noviembre 2017.
7. Levis S, Morzunov SP, Rowe JE, et al. Genetic diversity and epidemiology of hantaviruses in Argentina. J Infect Dis 1998; 177: 529-38.
8. Gonzalez-Ittig R, Salazar-Bravo J, Barquez R, Gardenal C. Phylogenetic relationships among species of the genus Oligoryzomys (Rodentia, Cricetidae) from Central and South America. Zool Scr 2010; 39: 511-26.
9. Rivera P, González-Ittig R, Rossi Fraire H, Levis S, Gardenal C. Molecular identification and phylogenetic relationships among the species of the genus Oligoryzomys (Rodentia, Cricetidae) present in Argentina, putative reservoirs of hantaviruses. Zool Scr 2007; 36: 231-9.
10. Levis S, García J, Pini N, et al. Hantavirus pulmonary syndrome in northwestern Argentina: circulation of Laguna Negra virus associated with Calomys callosus. Am J Trop Med Hyg 2004; 71: 658-63.
11. Rivera P, González-Ittig R, Gardenal C. Preferential host switching and its relation with Hantavirus diversification in South America. J Gen Virol 2015; 96: 2531-42.
12. Padula P, Martinez VP, Bellomo C, et al. Pathogenic hantaviruses, northeastern Argentina and eastern Paraguay. Emerg Infec Dis 2007; 13: 1211-4.
13. Levis S, Rowe J, Morzunov S, Enria D, St Jeor S. New hantaviruses causing hantavirus pulmonary syndrome in central Argentina. Lancet 1997; 349: 998-9.
14. Ksiazek T, Peters CJ, Rollin P, et al. Identification of a new North American hantavirus that causes acute pulmonary insufficiency. Am J Trop Med Hyg 1995; 52: 117-23.
15. Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposyum Series (Oxf) 1999; 41: 95-8.
16. Darriba D, Taboada GL, Doallo R, Posada D. J ModelTest 2: more models, new heuristics and parallel computing. Nat Methods 2012; 9: 772.
17. Huelsenbeck J, Ronquist F. MRBAYES: Bayesian inference of phylogenetic trees. BMC Bioinformatics 2001; 17: 754-5.
18. Calderón G, Pini N, Bolpe J, et al. Hantavirus reservoir hosts associated with peridomestic habitats in Argentina. Emerg Infec Dis 1999; 5: 792-7.
19. Mills J, Childs J, Ksiazek T, Peters CJ, Velleca WM. Métodos para trampeo y muestreo de pequeños mamíferos para estudio virológicos. Washington DC: Organización Panamericana de la Salud, 1998. Reporte No. OPS/HPS/HCT98.104.
20. Swofford, D.L. PAUP*.Phylogenetic Analysis Using Parsimony (* and Other Methods).Version 4. [Computer software and manual] Sunderland, Massachusetts: Sinauer Associates 1998. En: http://benedick.rutgers.edu/software-manuals/PAUP4-manual.pdf; consultado noviembre 2017.
21. Ciancaglini M, Bellomo CM, Torres Cabreros CL, et al. Hantavirus pulmonary syndrome in Tucumán province associated to an unexpected viral genotype. Medicina (B Aires) 2017; 77: 81-4.
22. Gonzalez-Ittig RE, Rivera PC, Levis SC, Calderón GE, Gardenal CN. The molecular phylogenetics of the genus Oligoryzomys (Rodentia: Cricetidae) clarifies rodent host-hantavirus associations. Zool J Linn Soc 2014; 171, 457-4.
